Cancer Gene Census(CGC)는 암과 관련된 돌연변이를 포함하는 유전자를 분류하고 이러한 유전자의 기능 장애가 어떻게 암을 유발하는지 설명하기 위해 지속적인 노력하는 데이터 입니다.
“지속적인 노력” 이라는 표현은 해당 유전자의 장애가 해당 암으로의 인과관계가 있다고 완벽히 설명되지 않기 때문인 것 같습니다.
하지만 새로운 증거가 밝혀지면 계속 업데이트 된다고 하고 “지속적인 노력”을 하고 있다는 것 같습니다.
Cancer Gene Census의 내용, 구조, 큐레이션 과정은 해당 논문에서 설명하고 있습니다.
A census of human cancer genes | Nature Reviews Cancer
자세한 내용은 논문을 살펴보면 될 것 같습니다.
현재 모든 인간 유전자의 1% 이상이 돌연변이를 통해 암과 관련되어 있다고 합니다.
(CGC 데이터를 보면 3% 이상인 것 같습니다만….)
이들 중 약 90%는 암의 체세포 돌연변이를 포함하고 있으며, 20%는 암에 걸리기 쉬운 생식세포 돌연변이를 갖고 있으며, 10%는 체세포 돌연변이와 생식세포 돌연변이를 모두 나타낸다고 합니다.
CGC는 암과 관련된 유전자를 증거수준에 따라 Tier 1과 Tier 2로 나누었습니다.
CGC의 Tier 구분은 ACMG Tier와 다릅니다. 오해가 없게 아래 설명을 참고해주세요.
Tier 1
Tier 1 유전자로 분류되려면, 암에서 해당 돌연변이가 유전자 산물의 활성을 변화시켜 암 형성을 촉진하는 증거가 있어야 합니다.
Cosmic CGC는 체세포 돌연변이 패턴도 고려합니다.
예를 들어, 종양 억제 유전자는 광범위한 inactivating 돌연변이를 나타내는 경우가 많으며, dominant oncogene들은 일반적으로 잘 알려진 hotspot에서 미스센스 돌연변이로 나타납니다.
oncogenic fusions에 관련된 유전자는 융합으로 인해 기능이 변경되어 발암성 변형이 발생하거나 (ex. dimerisation domain) 파트너에 조절 요소를 제공하는 경우(예: active promoter) Tier 1에 포함됩니다.
Tier 2
암에서의 역할에 대한 강력한 징후가 있지만 이용 가능한 증거가 적은 유전자로 구성됩니다.
이들은 일반적으로 그들의 역할을 뒷받침하는 증거가 계속 나오고 있는 보다 최근의 표적을 뜻합니다.
COSMIC CGC 둘러보기
우선 COSMIC CGC 파일을 불러옵니다. 733개의 로우로 이루어진 데이터 입니다.
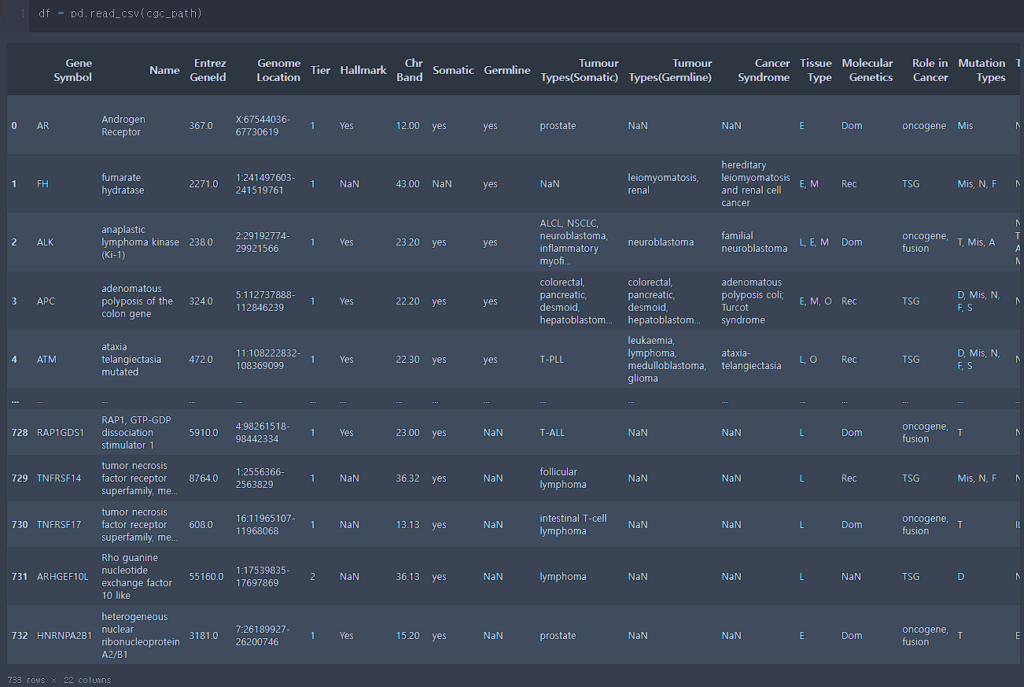
유전자도 733개인지 검사해봅니다.
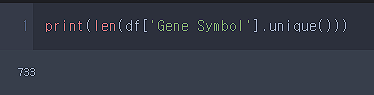
데이터 타입과 결측치 여부를 검사해봅니다
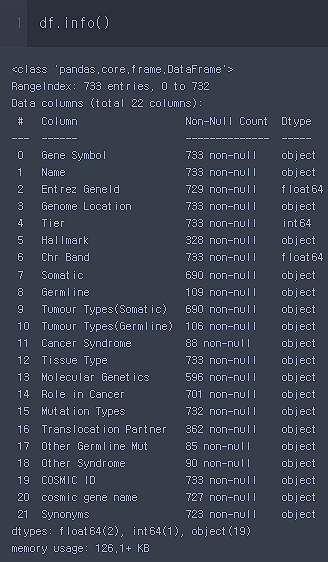
암과 관련된 유전자만 모은건데 암에서의 역할(Role in Cancer)가 701개 데이터만 있는 것을 확인 할 수 있습니다.
32개의 유전자는 암에서의 역할이 규정되지 않다는 의미입니다.
해당 데이터를 확인해 봅니다.
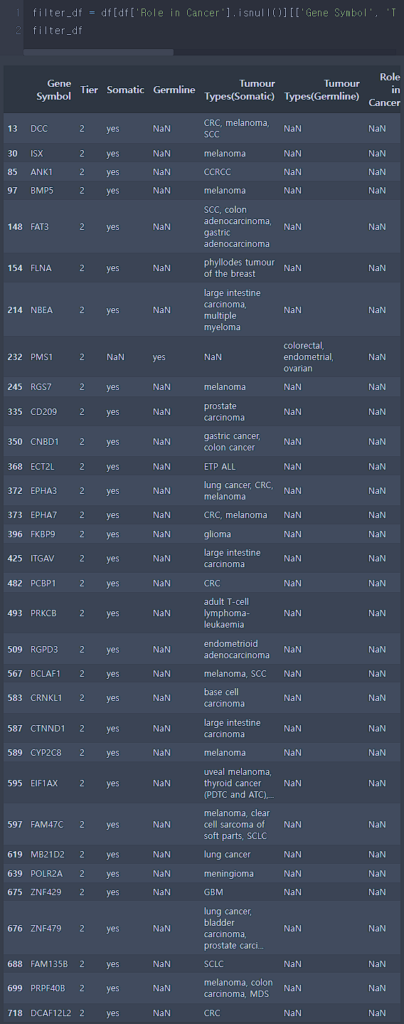
모두 Tier2에 해당하는 유전자인 것을 확인 할 수 있습니다. 암에서의 역할이 있다고 판단되지만 증거가 불충분한 유전자들 입니다.
PMS1만 germline mutation 유래로 인한 암을 유발한다고 합니다.
PMS1에 대해서 검색해봅니다.
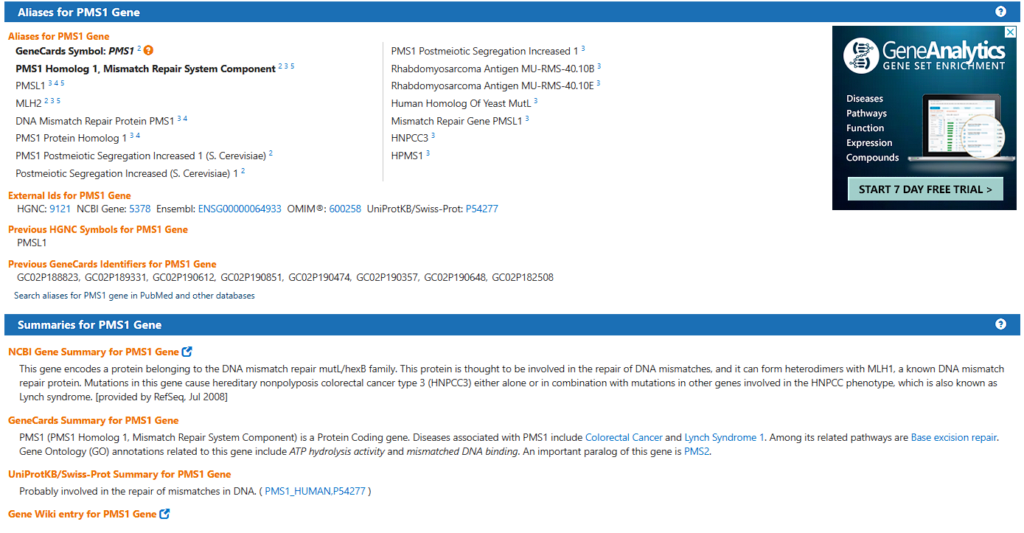
PMS1은 Mismatch repair protein이네요. Tumor Suppressor Gene 인 것을 확인 할 수 있습니다.
나머지 유전자들도 어떤 기능을 하는지 궁금해집니다. 하나 하나 검색하기 힘들기 때문에 Entrez를 이용해보겠습니다.
Biopython의 Entrez를 사용하여 입력한 유전자들의 정보를 가져올 수 있습니다.
유전자 정보를 가져온 후. DataFrame에 넣는 함수까지 작성하였습니다.
from Bio import Entrez
def get_gene_summary(gene):
Entrez.email = 'quasar2yh@gmail.com'
handle = Entrez.esearch(db='gene', term=gene)
record = Entrez.read(handle)
handle.close()
# 검색 결과에서 유전자 ID 추출
id_list = record['IdList']
if len(id_list) > 0:
gene_id = id_list[0]
handle = Entrez.efetch(db='gene', id=gene_id, retmode='xml')
record = Entrez.read(handle)
handle.close()
# 유전자 정보 출력
return record[0]['Entrezgene_summary']
def get_gene_summary_tables(gene_list):
gene_dic_for_df = {}
for gene in gene_list:
try:
gene_summary = get_gene_summary(gene)
gene_dic_for_df[gene] = [gene_summary]
except TypeError:
print(gene, len(gene_summary))
gene_df = pd.DataFrame(gene_dic_for_df).T
gene_df.rename(columns={0:'summary'}, inplace=True)
return gene_df
gene_df = get_gene_summary_tables(filter_df['Gene Symbol'])궁금했던 유전자 정보를 추출하였습니다.
그런데 summary가 너무 길이서 생략된 것을 확인 할 수 있습니다.
이런 경우 Excel이나 TSV 파일로 변환하여 확인 할 수도 있지만 주피터 노트북의 환경을 변경하여 내용을 확인 할 수도 있습니다.

아래 코드를 실행하면 주피터 노트북 환경에서 셀 내의 내용이 길더라도 모두 표기되도록 변경할 수 있습니다.
# Cell의 모든 내용이 표시되도록 설정 해줍니다.
pd.set_option('display.max_colwidth', None)
gene_df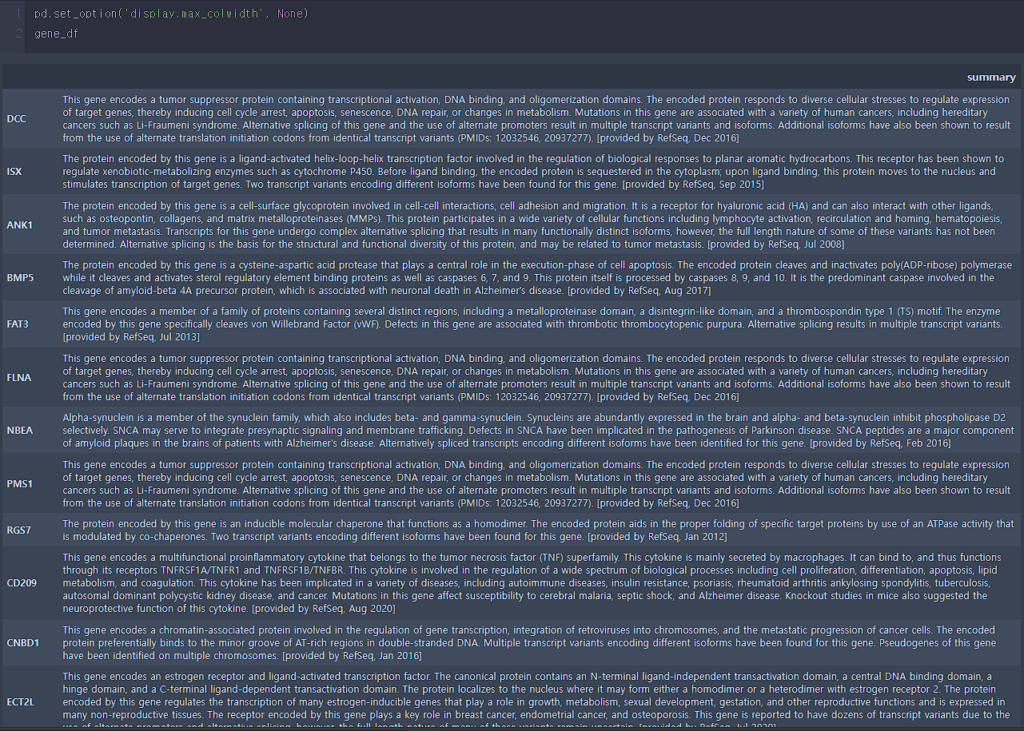
그림이 잘리긴 했지만 모든 내용을 한 눈에 볼 수 있도록 출력 된 것을 확인 할 수 있습니다.
지금까지 정말 간략하게 CGC 데이터를 둘러보았습니다.
사용자에 따라서 원하는 정보를 뽑을 수 있을 것 같습니다.
