대학원을 다니던 2016년에는 CRISPR/Cas9을 이용하여 동물 모델링을 열심히 하고 있었습니다. CRISPR/Cas9를 engineering해서 염기만 바꾸는 TARGET-AID라는 것도 나오고 수 많은 논문이 나왔었는데 이번엔 또 Cpf1 이라는 녀석이 혜성처럼 나타나 난리였던 기억이 있습니다.
이번 글에서는 그 당시 논문과 리뷰를 읽고 정리했던 내용 중 Cas9 과 Cpf1 차이에 대해서만 간략하게 정리하여 포스팅 하였습니다.
아래 그림을 보시면 Cpf1과 Cas9의 차이점을 한 눈에 아실 수 있습니다. PAM이 다르고, gRNA의 구조가 다르고, 절단 모양이 다른 것이 눈에 띕니다.
이러한 눈에 띄는 차이 외에 특이도와 효율성에도 약간의 차이가 보인다고 합니다.
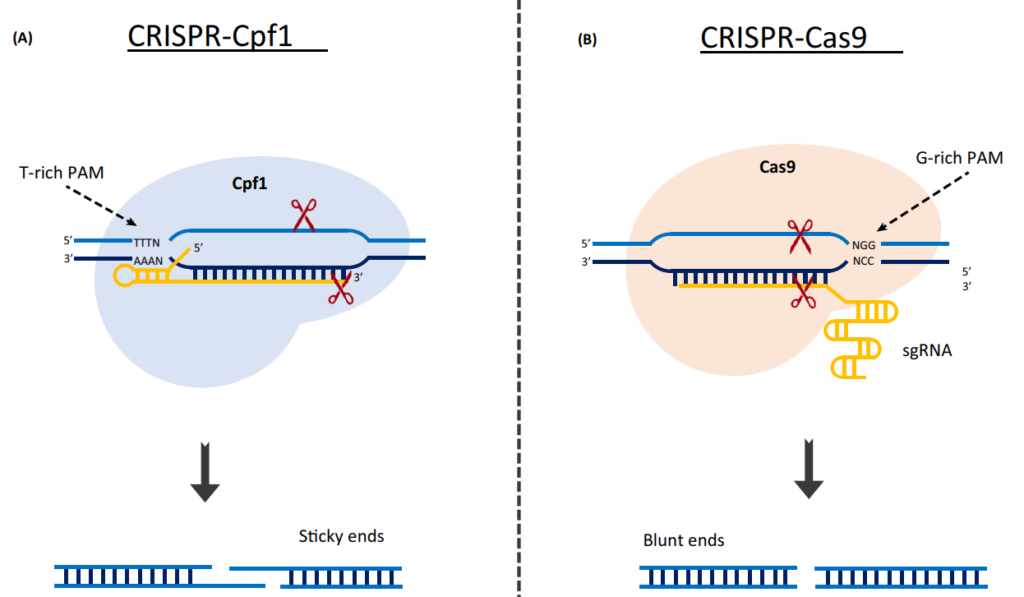
PAM 인식
Cpf1은 Protospacer Adjacent Motif (PAM)을 인식하는 방식이 SpCas9와 다릅니다.
SpCas9는 NGG (N = A/T/C/G) 를 인식하고 결합합니다.
반면에 Cpf1은 TTTV (T = Thymine, V = A/C/G) 또는 TTN PAM을 인식하고 결합합니다.
이러한 차이는 유전자 편집의 가능한 목표 DNA 위치를 다양화 시킵니다.
절단 위치
Cpf1은 target DNA에서 SpCas9에 비해 더 안쪽에 절단(cleavage) 위치를 가집니다.
SpCas9는 target DNA에서 PAM 다음에 위치한 3개의 염기 앞을 절단 합니다.
하지만 Cpf1은 PAM 보다 다소 안쪽 위치에서 절단을 수행합니다.
gRNA 구조
Cpf1과 SpCas9는 gRNA (guide RNA) 구조가 다릅니다.
SpCas9의 gRNA는 tracrRNA와 crRNA의 이중 구조로 구성되어 있습니다.
반면에 Cpf1의 gRNA는 crRNA와 tracrRNA를 결합한 단일 분자로 구성되며, SpCas9와 비교하여 더 간단한 구조를 가지고 있습니다.
Cpf1의 이런 특징은 연구자로 하여금 유전자 가위를 조금 더 쉽게 만들어서 적용이 가능하게 만들어 줍니다.
DNA 절단 패턴
Cpf1과 SpCas9는 DNA 절단 패턴도 다릅니다.
SpCas9는 대상 DNA 서열 양쪽에 blunt end 절단 부위를 만듭니다.
Cpf1은 더 내부적인 절단 위치에 sticky end 형태의 절단 부위를 만듭니다.
특이도
Cpf1과 Cas9의 특이도를 직접 딱 부러지게 비교하기는 어렵습니다.
왜냐하면 Cas9의 경우 PAM (protospacer-adjacent motif)이 NGG이고, Cpf1은 PAM이 Thymine이 많은 부위이기 때문에 둘이 절단할 수 있는 부위가 다르기 때문입니다.
같은 부위를 잘라내야 서로 효율성, 특이도 등을 직접 비교할 수 있는데 서로 다른 위치를 자르니 직접 비교가 어려운 것입니다.
그럼에도 불구하고 AsCpf1과 LbCpf1 두 가지의 Cpf1의 경우 특히 사람 세포에서 매우 특이적으로 작동하는 것이 확인되었으며 off target이 거의 나타나지 않는다는 것이 확인되었습니다.
